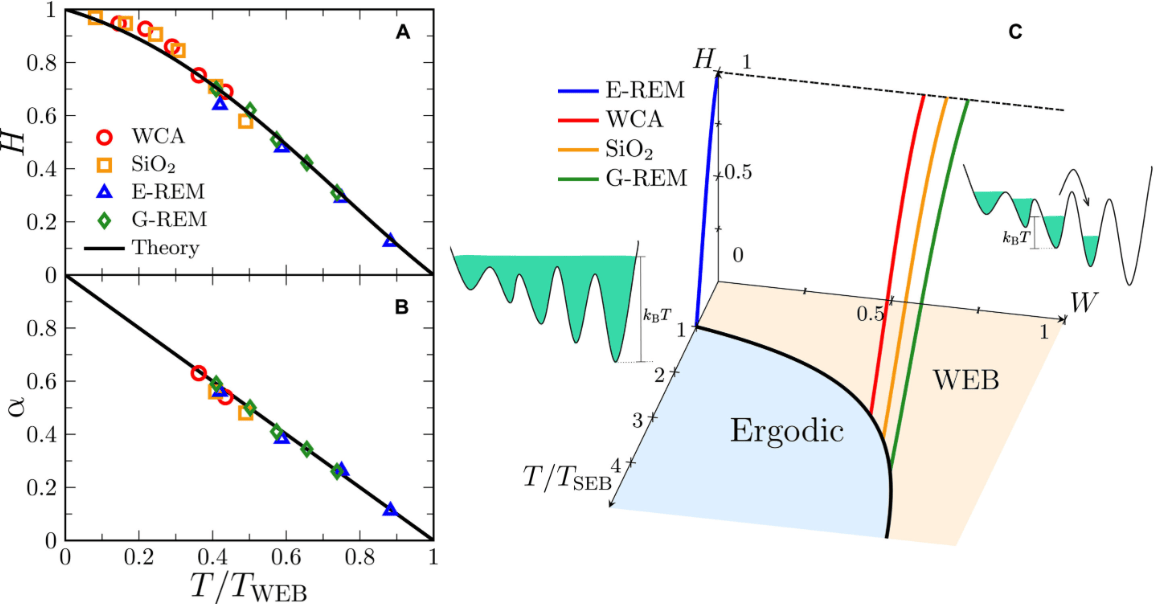
欧洲分子生物学实验室与乌普萨拉大学联合团队在《自然通讯》发表研究成果,首次通过整合结构生物学方法观察到RNA分子折叠的动态过程。这项RNA结构研究通过冷冻电镜、小角X射线散射及分子模拟等技术,记录了自剪接核酶从折叠组装到功能实现的完整过程。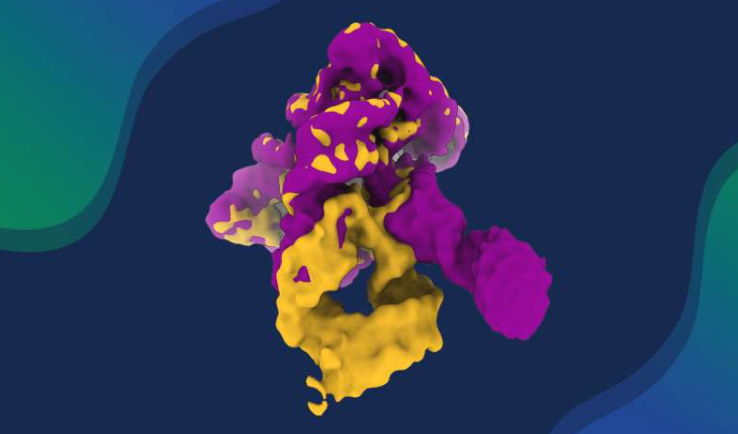
研究团队负责人Marco Marcia表示:"这项工作有望在塑造人工智能在RNA结构预测方面的应用方面发挥关键作用。"研究人员通过分析数十万单个RNA分子,重建了传统静态结构研究中难以捕捉的中间构象状态,揭示了RNA结构域按特定顺序组装的分子机制。
实验数据显示,结构域D1作为核心支架调控整个折叠过程,其构象变化引导其他结构域依次加入。曾任EMBL研究员的Shekhar Jadhav指出:"通过持续不断的努力和在电子显微镜下进行的大量筛选,我们最终得以观察到难以捉摸的RNA动态。"这种有序组装机制能有效避免RNA分子陷入错误折叠状态。
汉堡结构系统生物学中心开发的创新型图像处理方案,为捕捉RNA瞬态构象提供技术支撑。该中心教授Maya Topf表示:"这很好地展现了计算创新和高质量冷冻电镜数据如何揭示分子机器的隐藏构象。"结合分子动力学模拟,研究团队成功构建了RNA折叠过程的完整能量图谱。
这项RNA结构研究为理解生命进化提供新视角,第二类内含子核酶作为现代剪接体的祖先,其高效折叠机制揭示了早期RNA生命的演化路径。随着实验数据持续积累,这项研究将为人工智能预测RNA结构提供训练基准,推动RNA靶向药物与纳米生物技术领域发展。
更多信息: 利用冷冻电镜可视化大型多结构域核酶的动态组装,《自然通讯》(2025)。期刊信息: 《自然通讯》