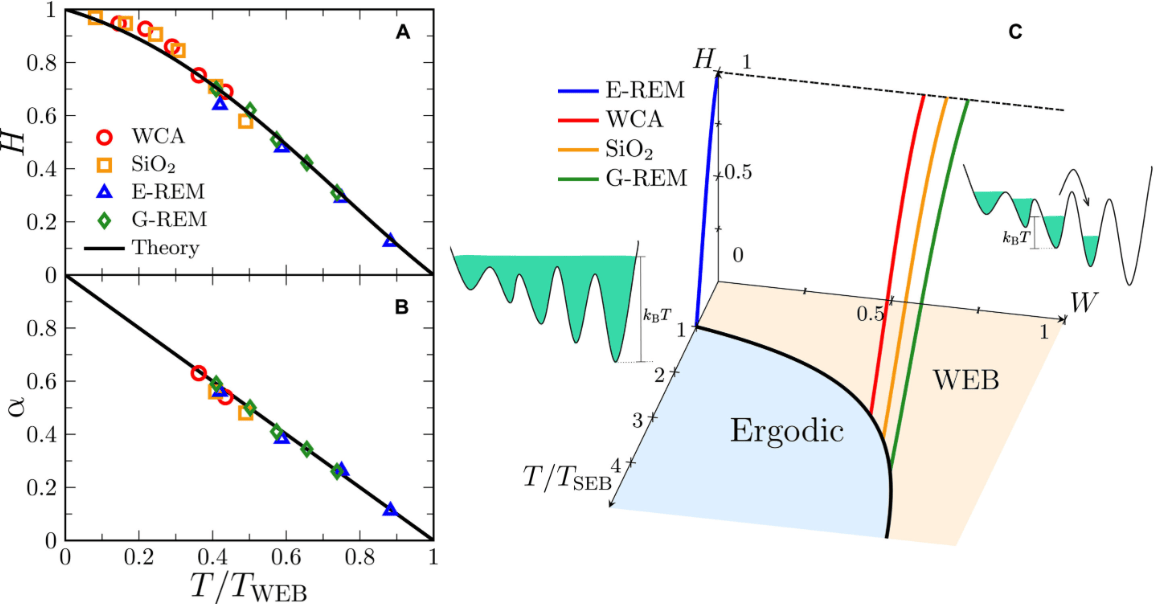
在豆科植物基因组学领域,中国科学院遗传与发育生物学研究所(IGDB)的研究人员取得了重要进展,成功为两种模式苜蓿物种制作了首个无间隙端粒到端粒(T2T)基因组组装。该研究由冯建博士和韩方朴博士领导,相关成果已发表在《分子植物》杂志上。
紫花苜蓿作为豆科植物生物学中的重要模式属,其根与根瘤菌共生形成的固氮根瘤对植物生长和土壤肥力提升具有关键作用。为深入了解这些性状的遗传和结构基础,完整准确的基因组组装必不可少。
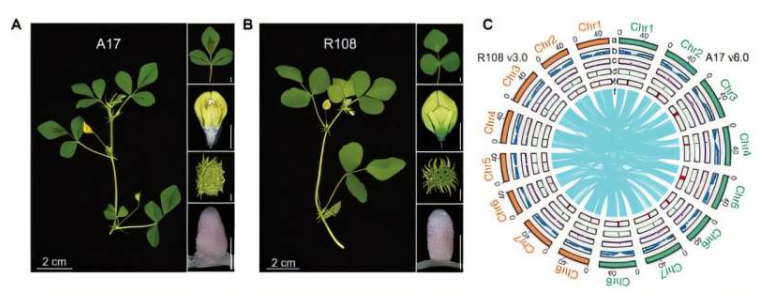
研究团队结合PacBio HiFi测序、超长Oxford Nanopore读长和Hi-C染色质构象捕获技术,成功构建了Medicago truncatula Jemalong A17(A17 v6.0)和Medicago littoralis R108(R108 v3.0)的T2T基因组组装。这些组装分别跨越494.47 Mb和415.27 Mb,完整性超过99%,代表了苜蓿属迄今为止最完整的基因组。
在解码着丝粒方面,研究人员取得了重要发现。着丝粒是真核生物基因组中最神秘的区域之一,在染色体分离中起核心作用,但由于其高度重复的特性,一直难以组装。通过整合多种技术手段,研究人员发现了A17和R108着丝粒之间的显著差异。A17着丝粒主要由两个串联卫星重复序列组成,而R108着丝粒则几乎完全由CentM168组成,这凸显了M. truncatula和M. littoralis之间独特的进化轨迹。
此外,研究人员还鉴定出几个高度染色体特异性的卫星重复序列,并发现着丝粒区域富含年轻的长末端重复序列(LTR)逆转录转座子,这表明转座因子在塑造着丝粒结构和功能方面可能发挥着重要作用。
该研究的通讯作者冯建博士表示,这项研究代表着对豆科植物基因组结构和着丝粒进化理解的一次重大飞跃,不仅为未来的基因发现和性状图谱提供了高质量的基因组基础,还为设计具有更高稳定性和传递保真度的植物染色体奠定了基础。共同通讯作者韩方朴博士也指出,有了完整的组装和详细的重复图谱,可以开始提出关于着丝粒如何起源、多样化以及如何影响豆科植物基因组进化的新问题。
据悉,这项研究中产生的T2T苜蓿基因组已公开,预计将加速豆科植物生物学的研究,涵盖从共生信号和压力适应到染色体工程和合成生物学等多个领域。
更多信息: Lisha Shen 等人,《两个完整的苜蓿端粒基因组揭示着丝粒的形态和进化》,《分子植物》(2025)。