莱布尼茨研究所(IPK Leibniz Institute)研究人员在植物育种全基因组预测领域取得新进展,相关研究成果发表于《植物生物技术杂志》。
受小麦杂交育种成功成果启发,研究人员将全基因组预测方法扩展至自交系,首次整合四个商业小麦育种项目的表型和基因型数据。近年来,深度学习方法在基因组预测中愈发重要,与传统方法相比,其能对输入数据进行灵活非线性变换,识别数据模式并与产量、株高等可观察特征关联,在植物特性受复杂相互作用影响时优势明显。
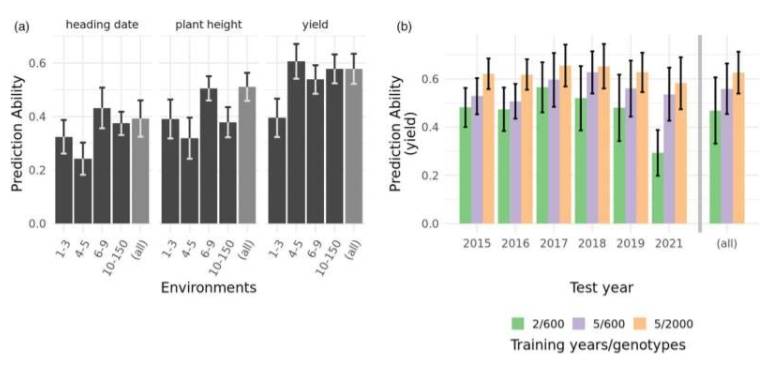
在此背景下,IPK研究团队承担学术数据受托人角色,将四个小麦育种项目数据与早期公私合作伙伴关系试验数据合并。IPK“育种研究”部门负责人Jochen Reif教授表示,研究需要不同环境(地点)测试过的多种基因型数据。新数据集涵盖168个环境中12年的试验活动,形成包含多达9500个基因型的基因组预测训练集,涉及谷物产量、株高和抽穗期。不过,合并不同数据并使其可比性成为一大挑战。
尽管表型和基因型信息存在差异,研究团队仍通过细致数据准备(包括缺失的SNP估算)打破公司数据库孤岛,获得可链接数据。随后,团队利用这些数据对比经典基因组预测方法与基于神经网络的深度学习方法,借助神经网络识别结构化数据模式。
研究第一作者Moritz Lell解释,分析表明不同测试序列可灵活组合用于基因组预测,且训练集规模增加(至少达约4000个基因型)时预测准确率会持续提升,但进一步增加训练集,预测值只会略有增加。对此,Reif教授强调,若在数据集中包含更多环境,有望克服这一瓶颈,更好地利用大数据在育种研究中的潜力。
更多信息: Moritz Lell 等人,《打破公司间数据孤岛,训练全基因组预测:小麦的可行性研究》,《植物生物技术杂志》(2025)。













 京公网安备 11010802043282号
京公网安备 11010802043282号