新南威尔士大学科学家团队开发出一种新型人工智能工具ECSFinder,该工具旨在解析人类基因组中占比98%的非编码区域。这项研究由新南威尔士大学与加拿大蒙特利尔大学、麦吉尔大学合作完成,研究成果已发表于《核酸研究》期刊。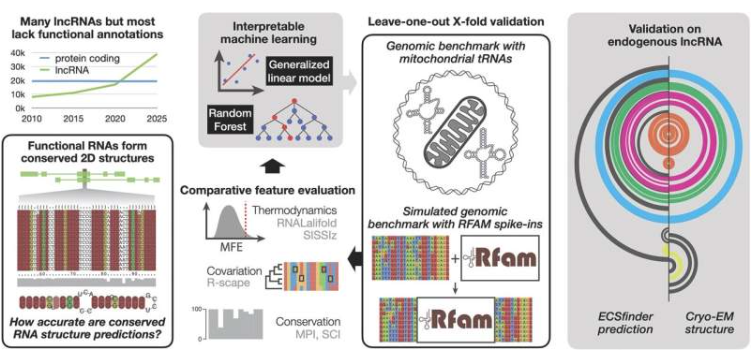
人类基因组中仅有约2%的序列负责编码蛋白质,其余部分曾长期被视为"垃圾DNA"。新南威尔士大学生物技术与生物分子科学学院副教授马丁·史密斯表示:"我们正在尝试解码人类基因组的逻辑电路,即告诉我们的DNA如何构建和运行人类的隐藏规则。"研究表明,许多疾病包括心脏病、癌症和某些精神疾病的根源可能位于这些非编码区域。
ECSFinder工具通过识别RNA结构来探索基因组功能。研究团队利用已知RNA结构训练人工智能系统,使其能够检测基因组中隐藏的调控元件。"我们期望发现数十万种新的RNA结构,从而为我们对基因组的理解增添新的维度,"史密斯副教授补充道。该工具在测试中表现出优于现有方法的性能。
基因组非编码区含有长链非编码RNA,这些分子具有关键调控功能,能激活或抑制特定基因表达。史密斯副教授解释说:"我们认为这些RNA就像软件一样,将蛋白质'硬件'协调成一个正常运转的交响乐。"这种调控机制的理解将为疾病治疗提供新思路。
研究人员指出,基因组非编码区的保守性特征表明其具有重要生物学功能。尽管人类与香蕉等生物在进化历程中分化,但部分基因组序列仍保持高度保守。ECSFinder工具的应用将有助于发现这些传统方法难以检测的调控元件。
该研究团队认为,对基因组非编码区的深入理解将推动个性化医疗发展。史密斯副教授表示:"由于RNA结构可以成为药物的靶点,它们为治疗提供了一个令人兴奋的新前沿。"通过解析个体基因组的非编码区域,未来可能针对特定疾病设计更精准的治疗方案。
更多信息: Vanda Gaonac'h-Lovejoy 等人,ECSFinder:从基因组序列优化预测进化保守的 RNA 二级结构,核酸研究(2025)。期刊信息: 核酸研究