格拉斯哥大学研究团队开发出新型蛋白质语言模型PLM-Interact,该模型能有效预测蛋白质相互作用并分析突变影响。这项发表于《自然通讯》的研究利用超级计算资源,为疾病机制研究和药物靶点发现提供了新工具。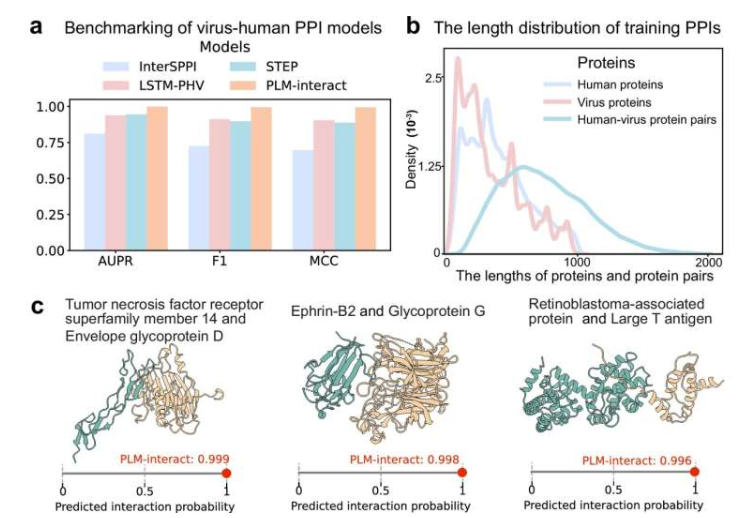
研究团队由癌症科学学院Ke Yuan博士、计算机科学学院Craig Macdonald教授和病毒研究中心David L Robertson教授领导,开发了基于大型语言模型的PLM-Interact蛋白质语言模型。该模型通过对421,000多对人类蛋白质相互作用数据的学习训练,展现出优于现有模型的预测性能。
Ke Yuan博士表示:"DiRAC超级计算机原本用于研究自然规律,现在帮助我们建立了探索蛋白质相互作用的新模型。计算机科学学院同事提供了语言建模支持,而DiRAC的计算资源使我们能更高效地完成这项工作。"该蛋白质语言模型在训练过程中涉及超过6.5亿个参数,利用英国DiRAC高性能计算设施的GPU集群加速模型构建。
PLM-Interact蛋白质语言模型在预测蛋白质相互作用方面的准确率较其他先进AI模型提升16%至28%。该模型成功预测了五种关键生物功能相关的蛋白质相互作用,而其他工具仅能预测其中一种。研究还证实该蛋白质语言模型能准确识别突变对蛋白质相互作用的影响,包括导致遗传疾病和癌症的突变类型。
研究人员使用22,383个来自人类和病毒蛋白质的相互作用数据进一步训练模型。PLM-Interact在预测病毒与宿主蛋白质相互作用方面同样表现优异,展示了其在病毒研究领域的应用潜力。David L Robertson教授指出:"COVID-19疫情期间凸显了理解病毒与宿主相互作用的紧迫性。PLM-Interact这样的工具能帮助我们更好理解病毒出现和疾病风险。"
这种蛋白质语言模型的开发为大规模精准预测蛋白质相互作用提供了新平台,未来将在疾病机制研究和治疗开发中发挥重要作用。
更多信息: Dan Liu 等,PLM-interact:扩展蛋白质语言模型以预测蛋白质-蛋白质相互作用,《自然通讯》(2025)。期刊信息: 《自然通讯》