4月16日,一项由我国中国科学院分子植物科学卓越创新中心韩斌教授团队领导的研究在线发表于《自然》杂志。该研究构建了前所未有的野生稻和栽培稻泛基因组图谱,深入解析了水稻的遗传结构和多样性,为水稻育种和农业创新提供了强大资源,也为水稻的进化和驯化历史带来了全新见解。
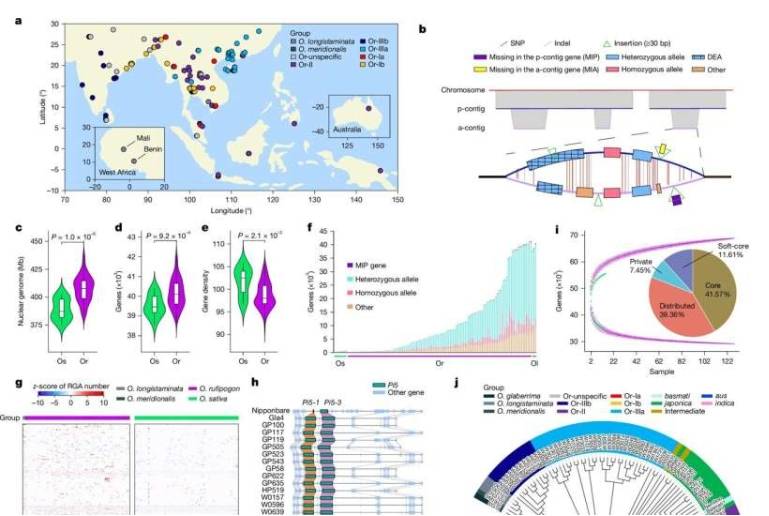
在全球气候变化和人口快速增长的双重压力下,作为全球数十亿人主食的亚洲栽培稻(Oryza sativa),迫切需要提升抗病虫害能力和产量。尽管驯化水稻已得到广泛研究,但其野生祖先——普通野生稻(Oryza rufipogon)因历经数千年适应多样化环境,在很大程度上仍未被充分研究,在理解野生稻全部遗传潜力方面存在关键空白。
此次研究中,研究人员获取了145个地理分布和遗传多样性丰富的水稻基因组,涵盖129个野生种质和16个栽培品种。借助先进的PacBio高保真(HiFi)测序技术和计算方法,构建了迄今分辨率最高的“泛基因组”,成功捕捉野生稻完整遗传图谱,揭示了对作物改良极为关键的隐藏变异。
研究发现了38.7亿个碱基对的新基因序列,这些序列在单一公认的参考基因组(O. sativa ssp. japonica cv. Nipponbare)中缺失,还有69,531个遍布整个泛基因组的基因。其中近20%的基因仅存在于野生稻中,许多与抗病性和环境适应性相关,堪称“基因金矿”,有助于培育能抵御病虫害和气候变化挑战的现代水稻品种。
基于高质量基因组序列,研究人员对亚洲栽培稻各群体早期关键驯化基因开展单倍型分析,证明所有驯化位点均源自粳稻祖先Or-IIIa。这一发现有力支持了亚洲栽培稻各群体经历单一初始驯化事件的假说,为长期科学争论提供了重要证据。
此外,研究人员发现南亚栽培稻群体间存在广泛基因流动,对新发现的亚种群intro-indica进行了分类,并成功绘制了水稻进化和驯化的综合路线图。同时,还探究了籼稻和粳稻这两个主要亚种间的遗传分化,鉴定出超过85万个单核苷酸多态性位点和1.3万个存在-缺失变异。结果表明,这些变异主要源于各自祖先的分化以及粳稻中存在更大的遗传瓶颈,为整合不同水稻亚种的有益基因开辟了新机遇。
近乎饱和的泛基因组数据集整合了宝贵的野生遗传资源,为农业研究人员和植物育种者提供了坚实基础。科学家可借此挖掘有利等位基因,追踪关键基因起源,加深对水稻环境适应性和表型可塑性的理解。该研究为培育能抵御极端气候、消耗更少资源且产量更高的水稻品种提供了路线图。
更多信息: Dongling Guo 等,《野生稻和栽培稻的泛基因组参考》,《自然》(2025)。